3.シミュレーション結果の解析¶
前回までで、水とイオンが入ったボックス内のタンパク質のMDシミュレーションを行った。 今回は得られた最終構造とトラジェクトリを用いて、いくつかの分析をしてみる。
トラジェクトリの修正
RMSD
回転半径
トラジェクトリの修正¶
まずシミュレーションが正しく終了し、トラジェクトリファイル(md_0_1.xtc)が生成していることを確認する。 トラジェクトリファイルはシミュレーション中の全原子の軌跡を記録したファイルであり、GROMACSにはtrrファイルとxtcファイルの形式がある。
trrファイル : 原子の位置と速度の情報を含むバイナリファイル
xtcファイル : 原子の位置のみの情報を含むバイナリファイル
しかしながらVMDでトラジェクトリを確認すると、周期的境界条件により原子がボックスの反対側へジャンプするため、結合が正しく描画されない。 そこでgmx trjconvを使用して、トラジェクトリの座標や周期性を修正する。
| gmx trjconvオプション | 説明 |
|---|---|
| -s | 入力tprファイルの指定 |
| -f | 入力(修正前)トラジェクトリファイルの指定 |
| -o | 出力(修正後)トラジェクトリファイルの指定 |
| -pbc | 周期的境界条件の処理タイプの指定(molは分子の重心をボックス内に入れる) |
| -center | 指定したグループをボックス中心に配置 |
gmx trjconv -s md_0_1.tpr -f md_0_1.xtc -o md_0_1_noPBC.xtc -pbc mol -center
コマンドを実行すると中心に配置するグループを聞かれるので1(Protein)を選択し、さらにトラジェクトリを出力するグループとして0(System)を選択する。 修正後の「md_0_1_noPBC.xtc」をVMDで確認すると、先ほどまで見えていた結合の線が消え、見やすいトラジェクトリとなっていることがわかる。

Before : md_0_1.xtc

After : md_0_1_noPBC.xtc
これでトラジェクトリファイルの修正は完了である。 今回は1 nsのシミュレーションを行っており、10 psごとに座標を出力している。VMDで動きを確認してみよう。

RMSD¶
RMSDは平均二乗偏差(Root Mean Square Deviation)のことで、タンパク質の構造の非類似性を定量化したものである。 ここではタンパク質のダイナミクスにおいて、対応する各原子のずれの平均二乗偏差を考え、タンパク質の構造安定性を議論する。 修正したトラジェクトリファイルを用いて、gmx rmsによりRMSDを計算する。
| gmx rmsオプション | 説明 |
|---|---|
| -s | 入力tprファイルの指定(ここでの構造が参照される) |
| -f | 入力トラジェクトリファイルの指定(ここでの構造と比較する) |
| -o | 出力ファイルの指定 |
| -tu | 時間単位の指定(デフォルトはps、今回はns) |
gmx rms -s md_0_1.tpr -f md_0_1_noPBC.xtc -o rmsd.xvg -tu ns
最小二乗法とRMSD計算の対象グループに4(Backbone)を選択すると、平衡化されたタンパク質の構造を参照して水溶液中でのタンパク質のRMSDが計算され、rmsd.xvgが出力される。 同様にして、結晶状態(ダイナミクスを行う前)のタンパク質の構造を参照して、RMSDを計算してみる。
gmx rms -s em.tpr -f md_0_1_noPBC.xtc -o rmsd_xtal.xvg -tu ns
rmsd_xtal.xvgが得られたら、両者を重ねてグラフにプロットしてみる。
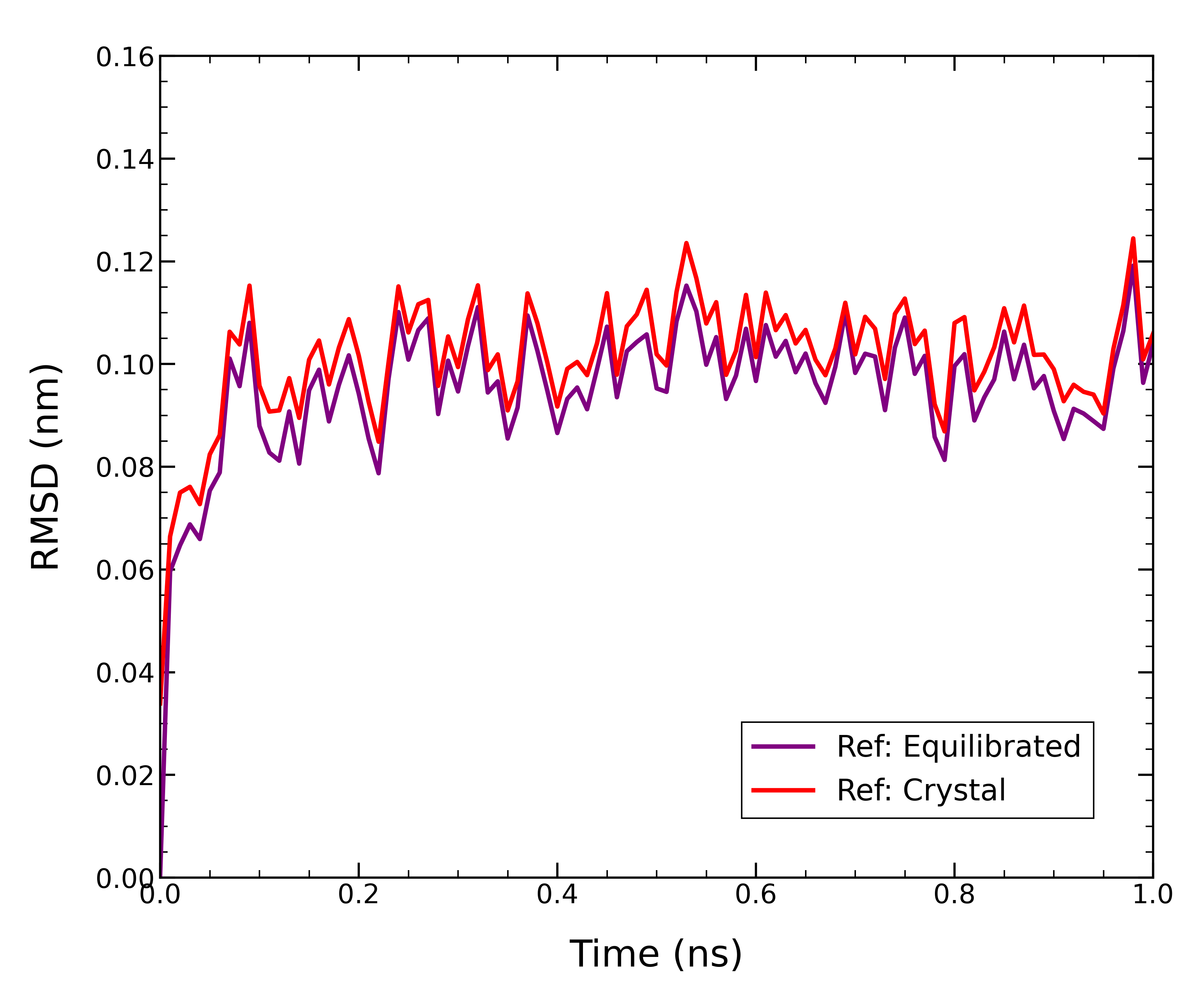
どちらのRMSDも約0.1 nm(1 Å)まで低下しており、タンパク質の構造が十分に安定化していることを示している。 また、プロット間の微妙な違いは平衡化されたタンパク質の構造が結晶状態の構造とわずかに異なることに起因している。 これは平衡化の過程での位置拘束が完全なものではないことや、エネルギー最小化された構造が必ずしも安定構造ではないことから予想される。
回転半径¶
タンパク質の回転半径Rgは、そのタンパク質がどのくらいコンパクトであるか(折り畳まれているか)を表す指標である。 タンパク質が安定に折り畳まれてる場合、Rgは比較的一定の値を維持する。 一方でタンパク質が展開した場合、Rgは時間とともに変化する値となる。 ここではgmx gyrateを用いて回転半径を計算する。
| gmx gyrateオプション | 説明 |
|---|---|
| -s | 入力tprファイルの指定 |
| -f | 入力トラジェクトリファイルの指定 |
| -o | 出力ファイルの指定 |
gmx gyrate -s md_0_1.tpr -f md_0_1_noPBC.xtc -o gyrate.xvg
回転半径の計算の対象グループとして1(Protein)を選択し、出力されたgyrate.xvgをグラフにプロットする。
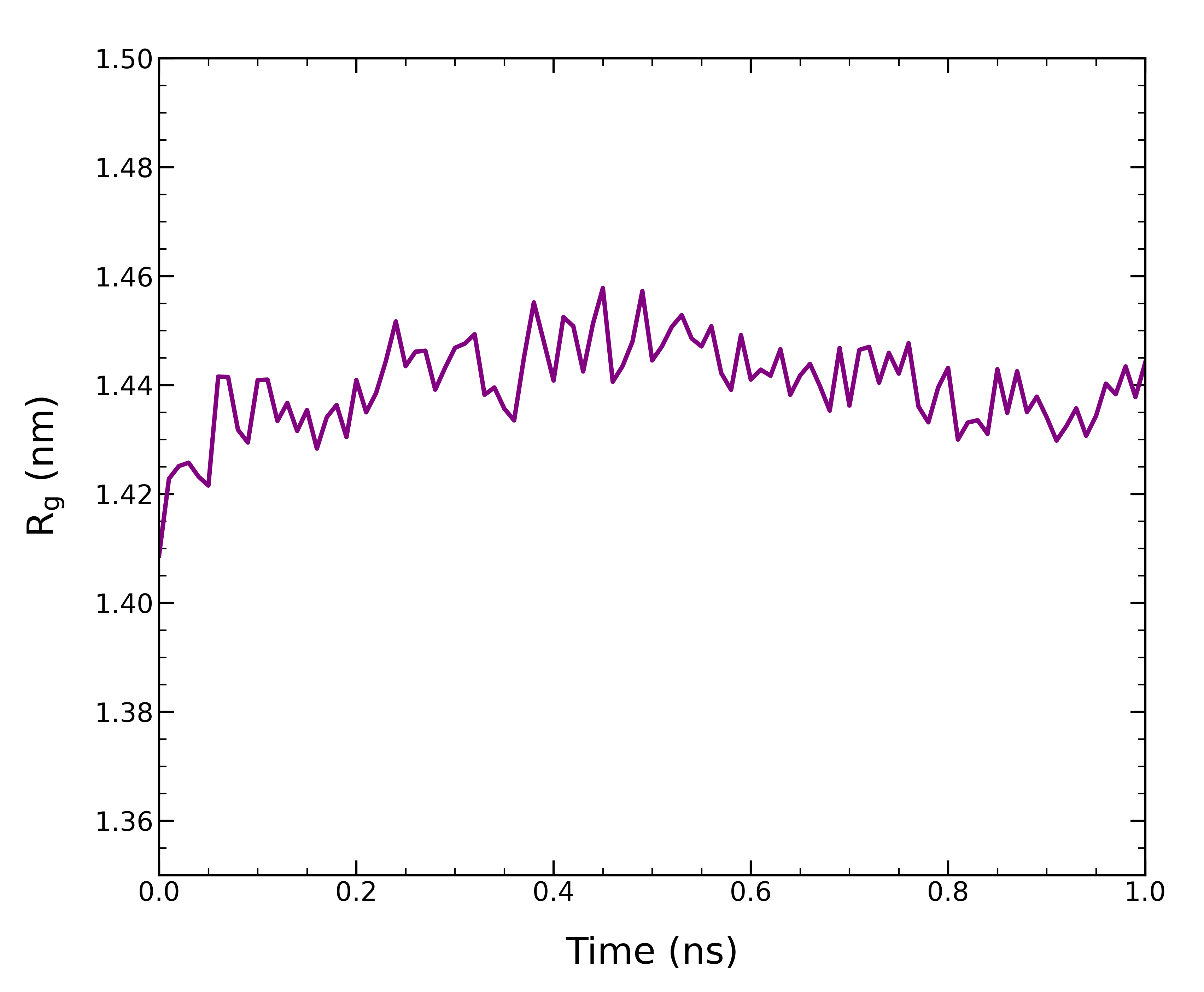
タンパク質の回転半径は300 Kで1 nsにわたってほぼ一定であり、タンパク質は折り畳まれた状態で安定していることがわかる。