2.MDシミュレーションの実行¶
前回、水とイオンの入ったボックス内にタンパク質が含まれる系を作成した。 今回はいよいよ、実際にMD計算を行っていく。
はじめに、MD計算の大まかな手順を解説する。 MD計算では系を作成した後、エネルギー最小化、平衡化を経てようやく本計算を行うことになる。 本計算の前に作成した系の構造を安定化しておくことで、シミュレーションをより現実の系に近づけることが可能となる。
エネルギー最小化
NVT平衡化
NPT平衡化
本計算(Production Run)
エネルギー最小化¶
系が作成できたら、まずはエネルギー最小化(Energy minimization)を行う。 これは、系に立体的な衝突や不適切なジオメトリがないように構造緩和を行う操作である。
エネルギー最小化のための入力ファイルはtprファイルであり、gmx grommpを使用して用意する。 エネルギー最小化における設定ファイルであるminim.mdpをここからダウンロードし、系の構造ファイル、トポロジーファイルとともにgmx grommpを実行する。
gmx grompp -f minim.mdp -c 1AKI_solv_ions.gro -p topol.top -o em.tpr
前回のイオンの追加ではtprファイルをgmx genionに渡したが、エネルギー最小化ではgmx mdrunを用いる。
| gmx mdrunオプション | 説明 |
|---|---|
| -v | 計算ステップを画面表示する |
| -deffnm | 入力ファイル名と出力ファイル名の指定 |
gmx mdrun -v -deffnm em
gmx mdrunの実行が終了すると、以下のファイルが得られる。
em.log : エネルギー最小化のログファイル
em.edr : バイナリのエネルギーファイル
em.trr : バイナリのトラジェクトリファイル
em.gro : エネルギー最小化が完了した構造ファイル
エネルギー最小化が成功しているかを判断するために、画面に出力されたポテンシャルエネルギーEpotと最大力Fmaxの値を確認しておく。
まず、ポテンシャルエネルギーEpotは負の値であり、系の大きさや水分子の数によるが、105から106のオーダーとなる。 また、最大力Fmaxは1000 kJ mol-1 nm-1以下の値となる。 これはmdpファイルにおいて、最大力がが1000 kJ mol-1 nm-1より小さくなるまでエネルギー最小化を行うように設定したためである。 mdpファイルの詳細については【mdpファイルの詳細】を参照してほしい。
ここで、エネルギー最小化におけるポテンシャルエネルギーの変化を調べてみる。 gmx energyを用いると、バイナリのエネルギーファイルem.edrからポテンシャルエネルギーのデータを取り出すことができる。
| gmx energyオプション | 説明 |
|---|---|
| -f | 入力edrファイルの指定 |
| -o | 出力ファイルの指定 |
gmx energy -f em.edr -o potential.xvg
対話形式で出力するエネルギーの種類を聞かれるので10(Potential)を入力し、終了のために0を追加で入力すると(10 0としてEnter)、画面にEtotの平均値が表示され、potential.xvgにポテンシャルエネルギーが書き込まれる。 potential.xvgをグラフにプロットすると、エネルギー最小化のステップに伴ってポテンシャルエネルギーが安定に収束していることを確認できる。

これにて系のエネルギーが最小化されたので、実際のダイナミクスに取り掛かる。
NVT平衡化¶
次のステップはNVT平衡化である。 実際のダイナミクスを行うためには、タンパク質周りの溶媒とイオンを平衡化しなければならない。 ここではNVTアンサンブルを適用して、300 Kで100 psの平衡化を行う。
NVT平衡化はエネルギー最小化と同様にgmx gromppとgmx mdrunを用いて行う。 NVT平衡化のgmx gromppに必要なmdpファイルはここからダウンロードする。
| gmx grommpオプション | 説明 |
|---|---|
| -r | 位置拘束パラメータの付与 |
gmx grompp -f nvt.mdp -c em.gro -r em.gro -p topol.top -o nvt.tpr
gmx mdrun -deffnm nvt
NVT平衡化におけるgmx gromppでは-rオプションに構造ファイルを指定することで、構造ファイルの座標に前回作成したposre.itpを適用し、タンパク質の重たい原子に対して位置拘束のパラメータを付与している。 これにより、タンパク質の構造変化を記述するための変数を追加することなく溶媒の平衡化を行うことができる。
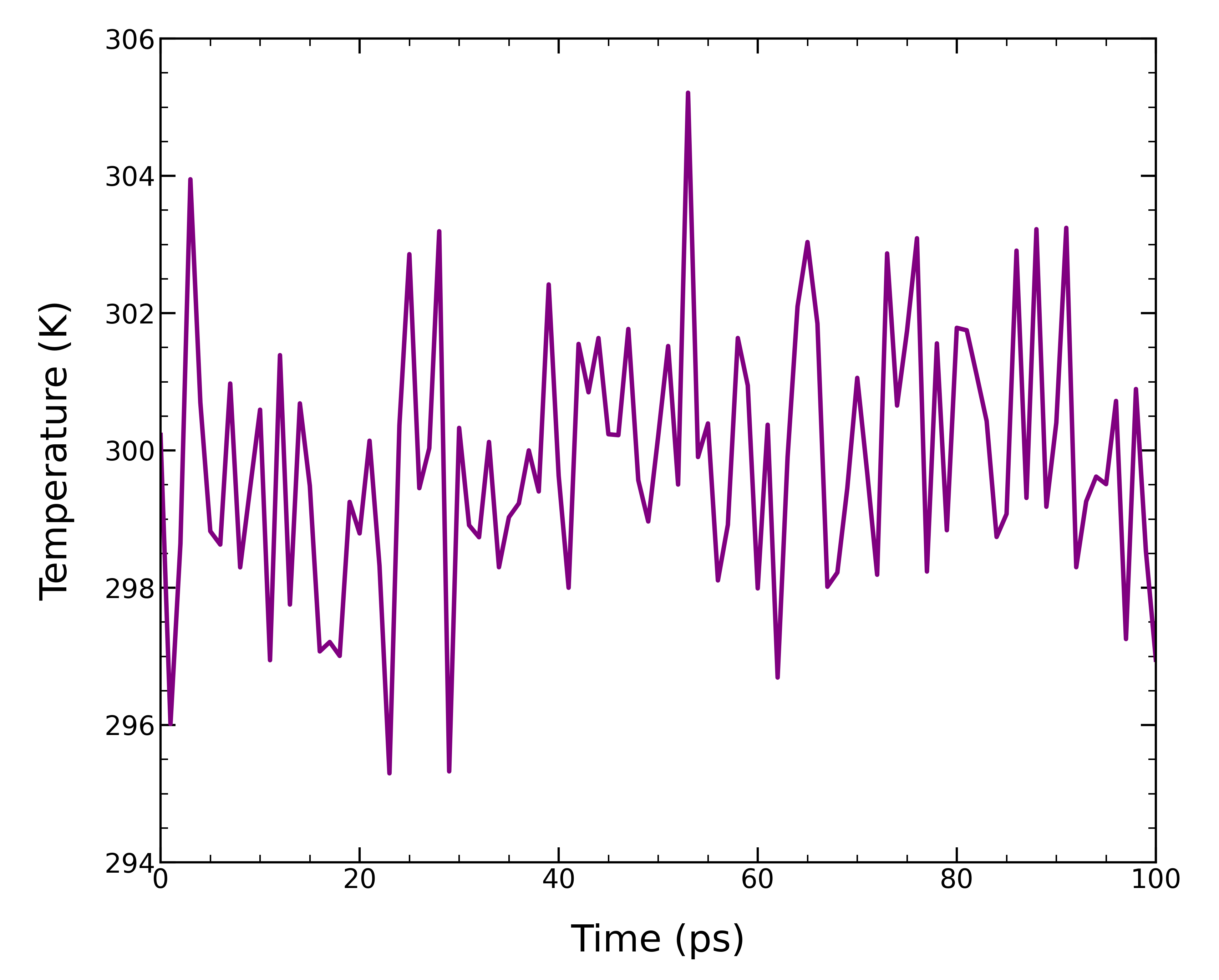
NVT平衡化が完了したら、系の温度制御が正しく行われていることを確認する。 gmx energyで16(Temperature)と0を選択してtemperature.xvgを出力する。
gmx energy -f nvt.edr -o temperature.xvg
グラフにプロットすると、系の温度がすぐに300 Kに達し、100 psの平衡化の間で安定していることを確認できる。
NPT平衡化¶
NVT平衡化により系の温度が安定したら、続いてNPT平衡化により圧力制御を行う。 NPTアンサンブルを用いて、300 K、1 barで100 psの平衡化を行う。
NVT平衡化と同様にgmx gromppとgmx mdrunを行う。 NPT平衡化のためのmdpファイルはここからダウンロードして使用する。
| gmx grommpオプション | 説明 |
|---|---|
| -t | チェックポイントファイルの指定 |
gmx grompp -f npt.mdp -c nvt.gro -r nvt.gro -t nvt.cpt -p topol.top -o npt.tpr
gmx mdrun -deffnm npt
ここでgmx gromppにおいて-tオプションを指定している。 これはNVT平衡化からシミュレーションを継続するためのチェックポイントファイル(cptファイル)を指定するものであり、NVT平衡化により生成した速度を引き継いでNPT平衡化を行うことができる。
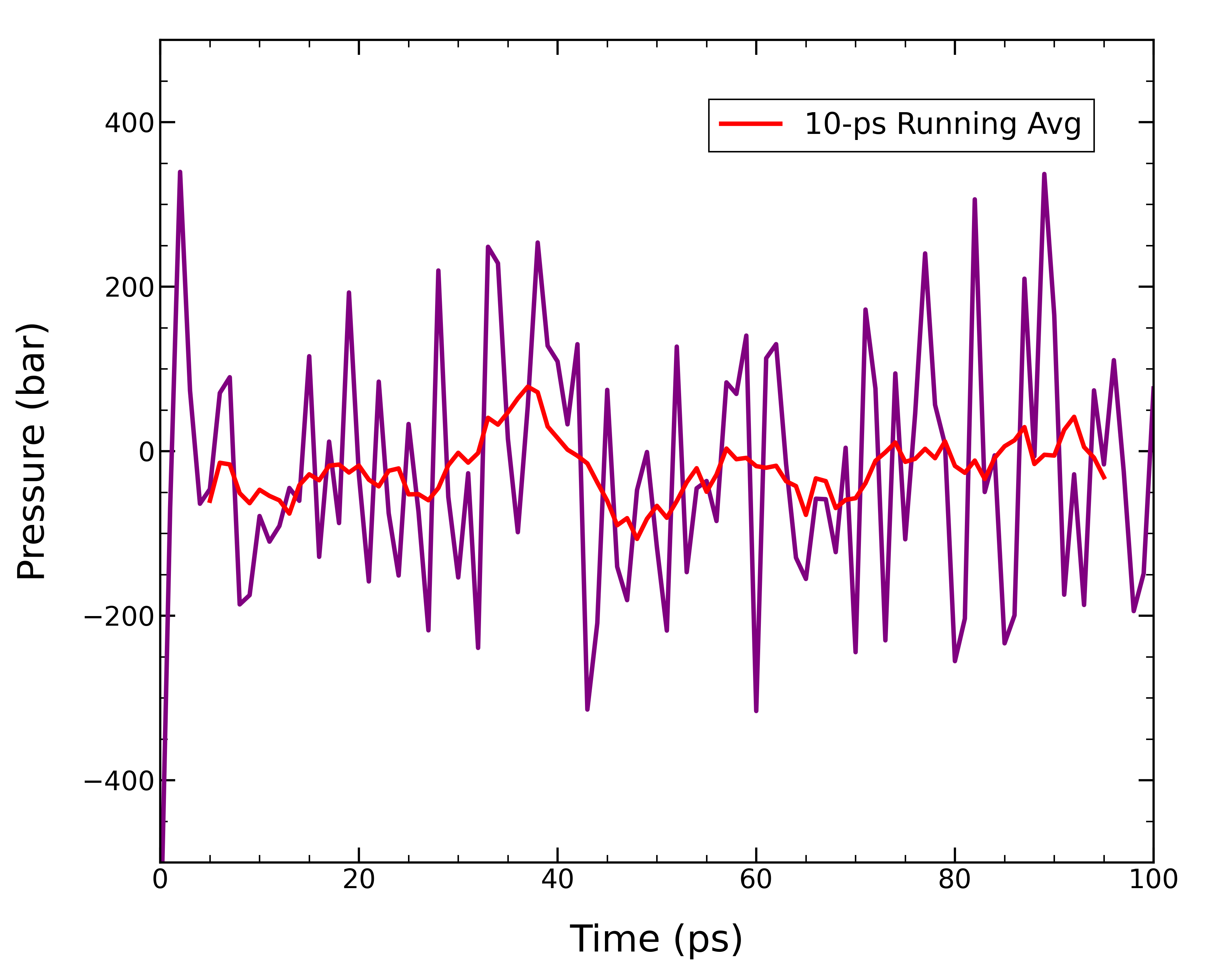
NPT平衡化が完了したら、今度は系の圧力制御が正しく行われていることを確認する。 gmx energyで18(Pressure)と0を選択してpressure.xvgを出力する。
gmx energy -f npt.edr -o pressure.xvg
グラフにプロットすると、圧力の変動はかなり大きいことが確認できる。 今回設定した基準圧力が1 barであるのに対して、平衡化での平均値は-25.6±157.9 barとなっている。 このように、圧力はMD計算において大きく変動する量であることを注記しておく。
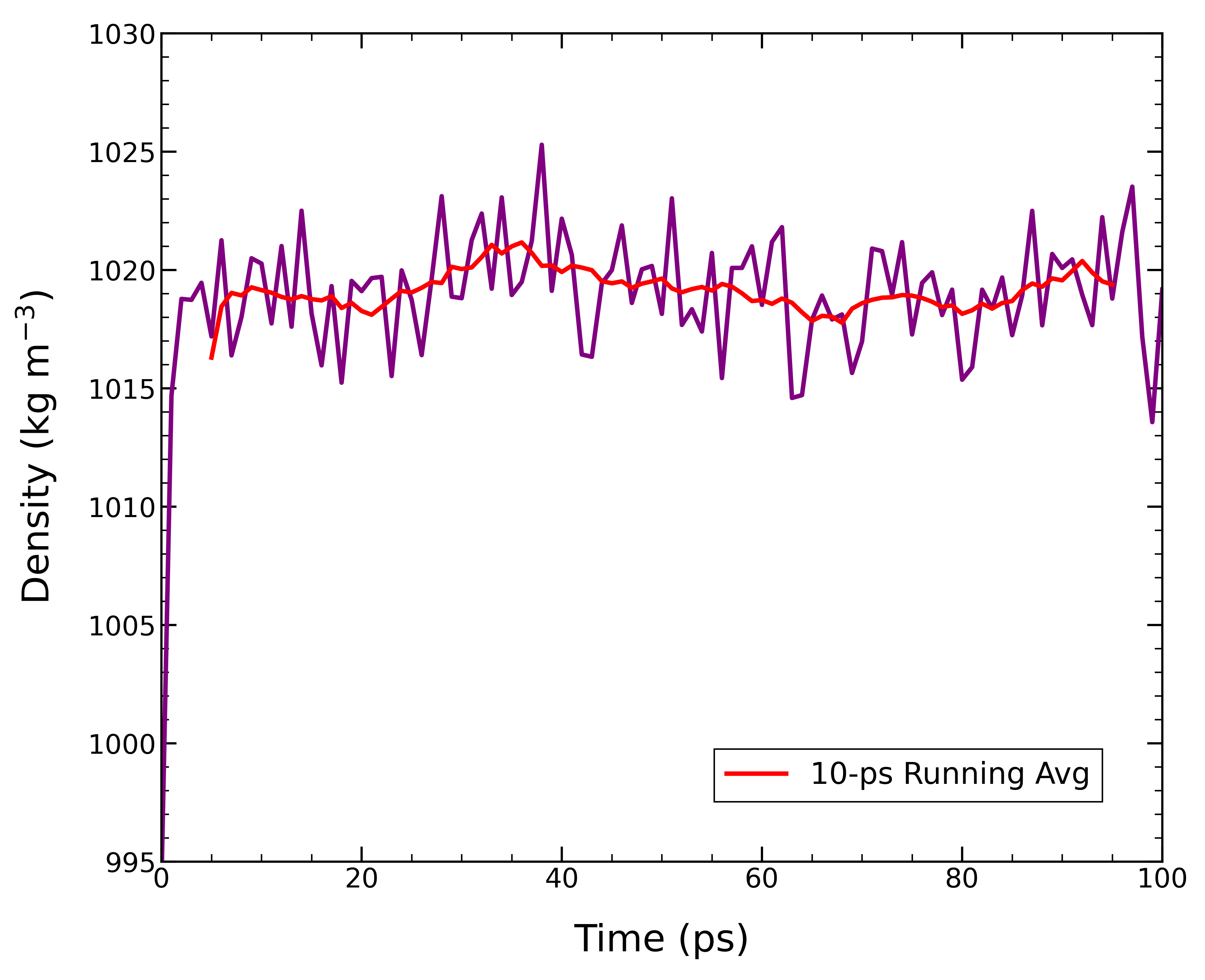
同様にして、密度変化も確認しておく。 gmx energyで24(Density)と0を選択し、出力されたdensity.xvgをグラフにプロットする。
gmx energy -f npt.edr -o density.xvg
密度の平均値は1018±3 kg m-3であり、実験値の1000 kg m-3やSPC/E水モデルの1008 kg m-3と近い値になっている。 密度の値は100 psの間非常に安定しており、これで系は圧力と密度に関して平衡化されたと判断することができる。
本計算(Production Run)¶
NVT平衡化、NPT平衡化のステップが完了すると、系は目的の温度と圧力で十分に平衡化されている。 ここからはようやく、シミュレーションデータを収集するための本計算(Production run)を行う。 今回は300 K、1 barを保ったまま1 nsのシミュレーションを行い、構造の変化とその軌跡をデータとして出力する。
-rで指定していた平衡化での位置拘束を外し、-tでNPT平衡化のチェックポイントファイルを指定してgmx gromppとgmx mdrunを行う。 本計算のためのmdpファイルはここからダウンロードして使用する。
gmx grompp -f md.mdp -c npt.gro -t npt.cpt -p topol.top -o md_0_1.tpr
gmx mdrun -deffnm md_0_1
計算が終了すると、最終的な構造ファイルと1 nsのトラジェクトリファイルが出力される。 以降はこれらのファイルを用いて、様々な解析を行うことになる。
まとめ¶
今回はMD計算の一連の流れを見てきた。手順をまとめておく。
エネルギー最小化により、系の構造緩和を行った。
NVT平衡化を行い、系の温度制御がなされていることを確認した。
NPT平衡化を行い、系の圧力制御がなされていることを確認した。
本計算を行い、解析に使用するシミュレーションデータを得た。
次回は得られたデータを用いた分析の例を示していく。