VMDについて2¶
VMD中級編¶
さて、前回まででVMDの簡単な使い方について説明した。
ここでは実際にVMDを使っていく上で実用的な知識を紹介する。
VMDの読み込み¶
初級編でvmd hoge.pdbとすればpdbを読み込めると説明した。トラジェクトリを読み込む場合も基本的には同様であるが、トラジェクトリファイルだけでは原子や残基の名前がないために上手く表示されない。そのためトラジェクトリを読み込む場合には
構造ファイル(pdb,psfなど) + トラジェクトリファイル(xtc,dcd,trrなど)
のようにして読み込む必要がある。
さらに例えばmd1.xtcに続くmd2.xtcを読み込みたい場合、
$ vmd hoge.pdb md1.xtc md2.xtc
とすれば読み込むことができる。
VMD 1.9.X OpenGL Display GUIの機能¶
上記の画像のGUIを以下ではDisplay GUIと呼ぶことにする。これまで初級編では主にGraphical Representations GUIの使い方を説明してきた。しかしDisplay GUIにもボタンはないが便利な機能が存在する。代表的なものを以下に載せておく。実際にVMDを立ち上げて試してみてほしい。
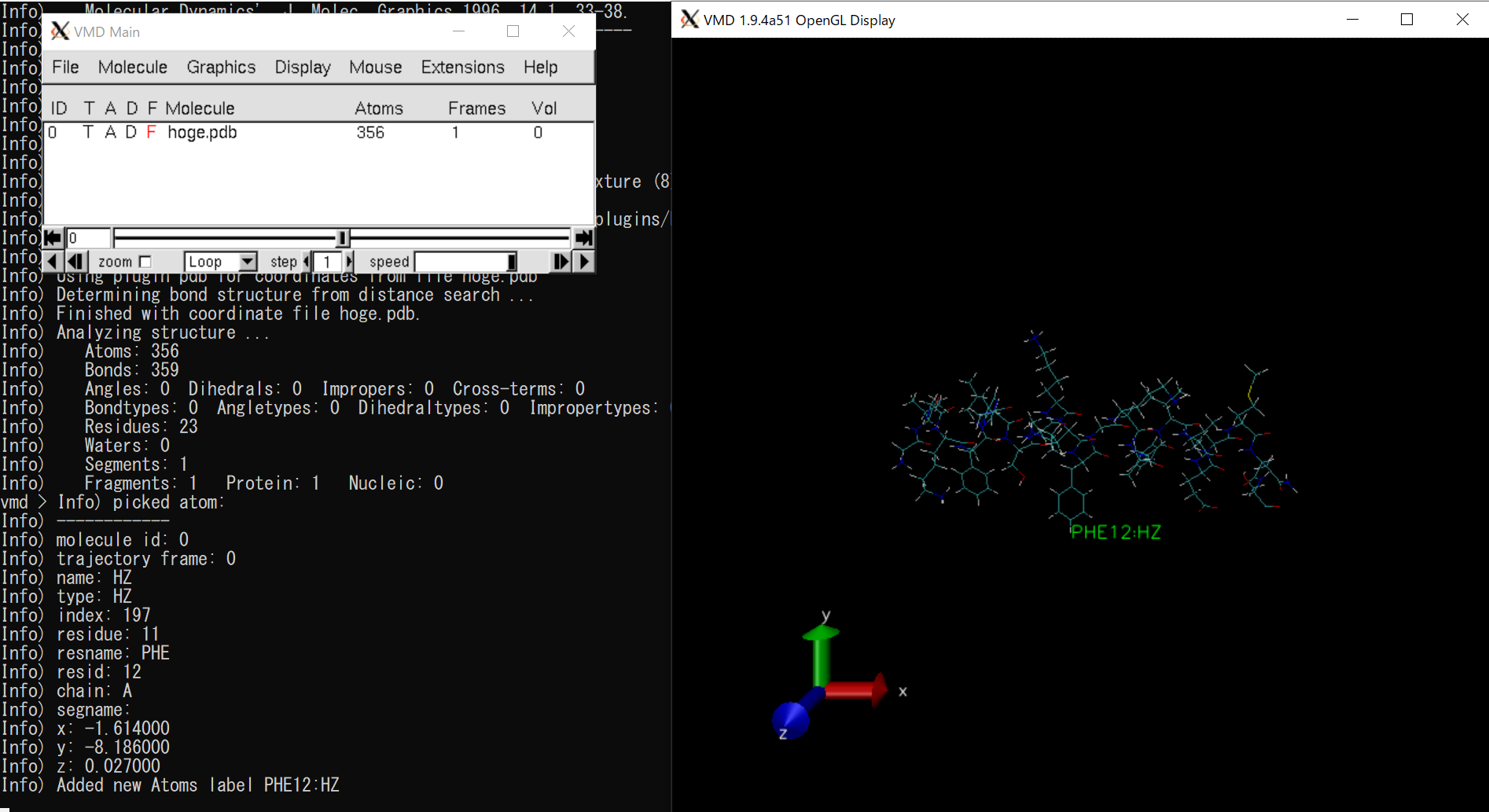
「1」キー を押してDisplay GUI上で原子をクリック: 原子の情報を表示。¶
「1」キーを押すとDisplay GUI上でのカーソルの形が変わる。その状態で原子をクリックすると上の画像のように選択原子の"resname"と"resid"及び"name"が表示される。さらに後ろのターミナルに注目してほしい。ターミナルにはindexやchain、またx,y,z座標といったさらに詳しい情報が表示される。
もし表示を消したい場合には再度同じ原子をクリックすることで表示が消える。
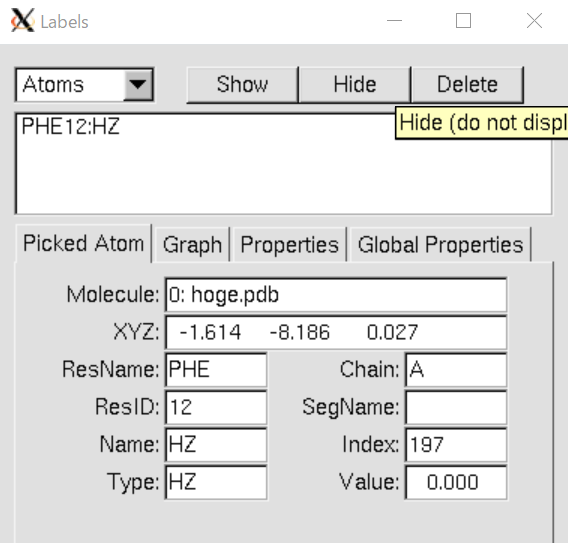
しかしターミナルはVMDのGUIと重なって見にくいという場合がある。その場合はVMD main GUIの"Graphics"タブ => "Labels..." の順番でクリックして欲しい。すると以下のようなGUIが新たに表示される。
ここにもターミナルに表示されている情報が全く同様に表示される。同じならターミナルで見ればいいという人もいるかもしれないがこのGUIを表示するメリットは他にもある。
先ほど原子情報の表示をやめたい時は同じ原子をクリックすればいいと述べたが、原子数が多い系では同じ原子を再度クリックすることは容易ではなく、中々表示を消せないどころか他の原子の情報を誤って表示してしまいかえって手間を増やすことになりかねない。そのような時にLabels GUIでPHE 12:HZと表示されている部分をクリックして選択し、その後**Hide**を押すことで非表示にできる。
また原子の選択項目をLabel GUI上から非表示にするだけでなく消したい場合はDeleteをクリックすればよい。
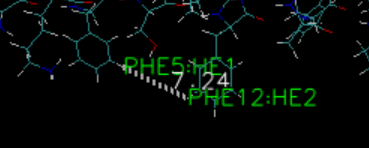
「2」キーを押してDisplay GUI上で原子を2つクリック: 原子間距離を表示¶
題の通り、[2]キーを押して距離を測りたい原子を2つクリックすることで原子間距離を表示することができる。
単位はÅで表示される。
「r」 キーを押してGUI上でドラッグ: 分子を回転¶
通常VMDを立ち上げた時はこのモードである。そのため「1」キーや「2」キーのモードから通常のモードに戻りたい時は「r」を押すことで戻せる。
「t」キーを押してGUI上でドラッグ: 分子を平行移動¶
系の端などに着目したい時は「t」キーを押してドラッグすることで視点を移動することができる。
「c」キーを押して原子をクリック: 回転中心を選択¶
VMDを立ち上げた際ドラッグして系を回転させる際の中心は系の重心になっているはずだ。その回転中心を変えたい時には「c」を押した後に回転中心にしたい原子をクリックすればよい。特定の残基や原子にズームして見たい時などに有効。
Graphical Representations GUI のより高度な原子選択¶
初級編では原子や残基といった分子の特定部分を指定する基本的な方法について紹介した。ここではより発展的な方法について紹介する。
Selected Atomsの文法¶
Graphical Representationsの"Selected Atoms"の指定ではプログラミング言語のように「かつ」、「または」、「でない(否定)」の文法が使用できる。例えば以下のように行う。
・「かつ」: chain A and resname GLY
・「または」: protein or lipid
・「でない」: not lipid
また必要に応じて「()」を使用することもできるが使用する機会は多くない。このページの「ある距離以内の原子の選択」の項目で「()」を使った例について取り上げるのでそちらの例を参考にしてほしい。
複数原子や残基の選択¶
上記文法を学んだからといって無闇に乱用するのは避けよう。例えば以下の例。
resname POPE or resname POPG or resname POPC
これはPOPE, POPG, POPCに含まれる全ての原子を表示するためのキーワードである。このような場合は次のように書くことができる。
resname POPE POPG POPC
また原子のindex(0始まりの番号)についてはさらに便利な指定がある。例えば5番から9番を選択したい時は
index 5 to 9
と書くことができる。residやserialでも同様にできるはずなので試してみてほしい。
ある距離以内の原子の選択¶
便利なキーワードに"within X of"がある。これは半径 X Åにある原子を選択する際に役立つ。利用例としては以下の通り。
water within 10 of (index 3626 to 3628)
少しこれまでの知識を活かした例を載せた。また先に述べた「()」を含んだ指定の仕方でもある。
意味としてはindex番号が3626から3628までの原子のそれぞれにおいて10Å以内にある水分子を表示するという意味である。原子全てを表示するとかえって見にくいということがあるのでこれにより必要な分子だけを表示できる。
※注意
上記の文法で分子を指定してトラジェクトリを再生するのは推奨しない。使い方としてはあくまでも特定の1フレームにおいて用いる使い方が好ましい。具体的例を出してその訳を説明する。
例えばion within 5 of (index 0 1)と指定したとしよう。そしてその際に実際に5Å以内にいたイオンのindexが3,4,5だったとしよう。この場合指定をしたフレームでは5Å以内にいたかもしれないが、次のフレームやその後のフレームでは5Å以内にいない可能性がある。にも関わらず、トラジェクトリを再生すると常にindex 3,4,5が表示され続ける。つまりトラジェクトリの各フレームにおいて5Å以内にいるイオンを表示するのは不可能であるということである。 代わりにその指定を行ったフレームのみでこの文法は有効であり、次のフレームに移った時に新たに5Å以内にいるイオンを読み込み直すというような操作は行われないということである。
この点を知らずにトラジェクトリを再生すると別のものを見ているということになりかねないので注意が必要である。
ある座標より大きいor小さい原子の指定¶
"Selected Atoms"では不等号も使用が可能である。具体的には以下のように使用する。
water and z<10
x < 15 or x>30
こちらはVMDで見るというよりは、初期構造の作製によく用いる。初期構造の作製の仕方は上級編で説明するのでそちらを参考にしてほしい。
分子が条件で分断されないように選択¶
こちらも上級編でまた改めて説明するが、先にこちらでも説明をしておく。例えば1つ上の項目で学んだようにx < 15 or x > 30としたとする。この場合もしとある水分子のH原子がx=14にあり、O原子がx=15にあるとH原子のみが表示される。表示だけなら大した問題ではないかもしれないが、構造を作る際には分子が切断されることになるため致命的な問題となる。このような場合には以下のようにする。
same residue as x < 15 or x > 30
こうすることで同一の"residue"として見なされる分子や残基が指定した境界(x=15やx=30)を跨いでいる際、その境界を超えていても切断されないように表示してくれる。つまりx=15にあるO原子も表示されるようになるということである。
構造を作る際にはかなり重要であるのでぜひ知っておいて欲しい。
まとめ¶
ここまで読んでいればVMDの扱いには基本的には困らないレベルにはなっているかと思う。
他にも筆者の知らない機能などはまだまだあると思うので、ぜひ調べて使ってみてほしい。
次のページではコマンドを用いた構造の作り方などさらに高いレベルのVMDの使い方を紹介する。
初めは難しいかもしれないが多くの人にとっては知っておくべき知識であると思うのでぜひ読んで欲しい。