VMDについて1¶
VMDのインストール¶
VMDのサイトからソースコードを取ってくる。
windows、mac、Linux用の中から自分のOSに適したものを選ぶ。筆者は1.9.3を使用しており、以下の名前のソースコードをダウンロードした。尚、ダウンロードにはアカウントの登録が必要である。
vmd-1.9.3.bin.LINUXAMD64-CUDA8-OptiX4-OSPRay111p1.opengl.tar.gz
インストールしたいディレクトリに圧縮ファイルを置き、
$ tar xvf vmd-1.9.3.bin.LINUXAMD64-CUDA8-OptiX4-OSPRay111p1.opengl.tar.gz
$ cd vmd-1.9.3
$ vi configure
#configureの中の$install_bin_dir="/usr/local/bin"と
#$install_library_dir="/usr/local/lib/$install_name"をそれぞれ自分の環境に直す。
#例えばbinもlibもvmd-1.9.3の中にあるため筆者の場合は
#$install_bin_dir="/home/hogehoge/apl/vmd-1.9.3/bin"とした。
#libについても同様
$ ./configure #./configureとすると以下のように出力される。
using configure.options: LINUXAMD64 OPENGL OPENGLPBUFFER FLTK TK ACTC CUDA IMD LIBSBALL XINERAMA XINPUT LIBOPTIX LIBOSPRAY LIBTACHYON VRPN NETCDF COLVARS TCL PYTHON PTHREADS NUMPY SILENT ICC
#そのためこのオプションをつけてconfigureを実行する。
$ ./configure LINUXAMD64 OPENGL OPENGLPBUFFER FLTK TK ACTC CUDA IMD LIBSBALL XINERAMA XINPUT LIBOPTIX LIBOSPRAY LIBTACHYON VRPN NETCDF COLVARS TCL PYTHON PTHREADS NUMPY SILENT ICC
$ cd src
$ make install
Make sure /home/kawabata/apl/tmp/vmd-1.9.3/bin/vmd is in your path.
VMD installation complete. Enjoy!
#上のような出力が出ていればインストールは成功している。
#あとはbinにパスを通してvmdコマンドを実行してみよう。
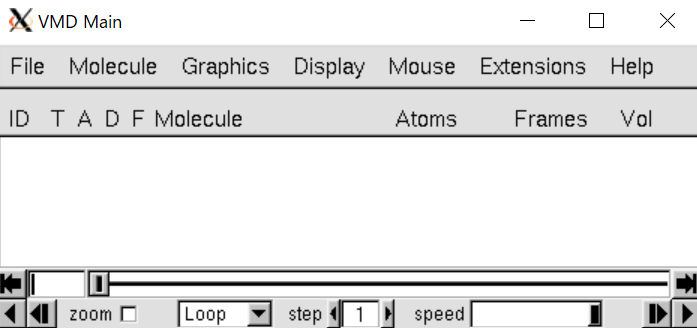
上記の結果次のようなGUIが立ち上がればインストールは無事終了である。
VMD初級編¶
vmdを実行すると上記の画像と共に下のようなGUIが表示されているはずだ。
このGUIは基本的にトラジェクトリの再生に使用する。分子を読み込んでいれば読み込んだ構造ファイルが表示される。試しに読み込んでみよう。読み込み方は2通りある。
コマンドで
vmd hoge.pdbなどとする。コマンドで
vmdとして先のように分子を読み込まずにVMDのGUIを立ち上げたあと、左上のFileタブを押す。その後New Molecule=>Browse...を押して目的のファイルをクリック、OKを押したあとLoadを押す。
1の方法の方が圧倒的に早く楽なため、基本的にこちらのやり方でロードするのが普通である。
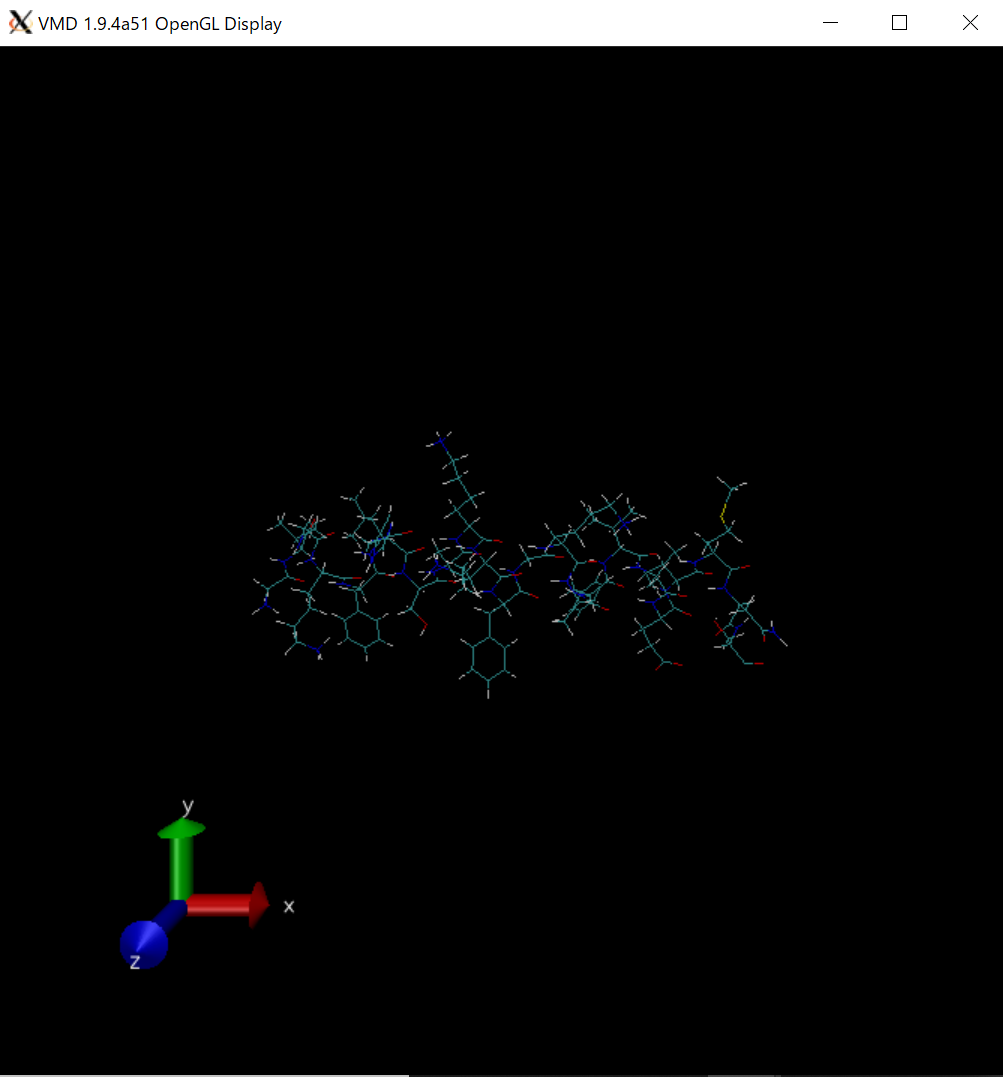
読み込むと以下のようになる。筆者はタンパク質を読み込んだ。
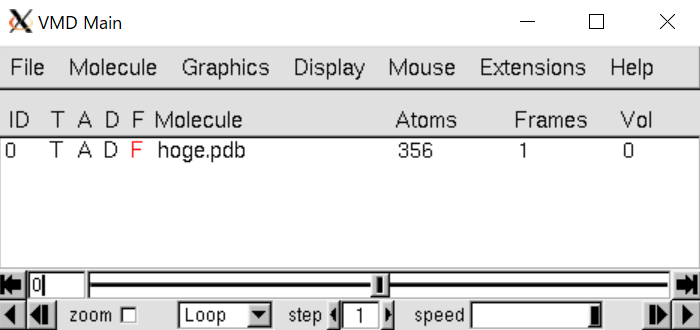
さて読み込むと"VMD Main"のGUIが変化した。
"Molecule"のところには読み込んだ構造ファイルの名前が記されている。
また一番左の"ID"はMolecule IDと呼ばれるものであり、VMDのコマンド(詳しくは上級編を参照)を使用する際に必要となるがここでは詳しくは述べない。
"Frames"はその名の通り何フレーム読み込んでいるかを表す。現在は構造ファイルの静止画を1つ読み込んだだけであるので1と表示されているが、トラジェクトリを読み込む場合はそのトラジェクトリのフレーム分の数値が表示される。
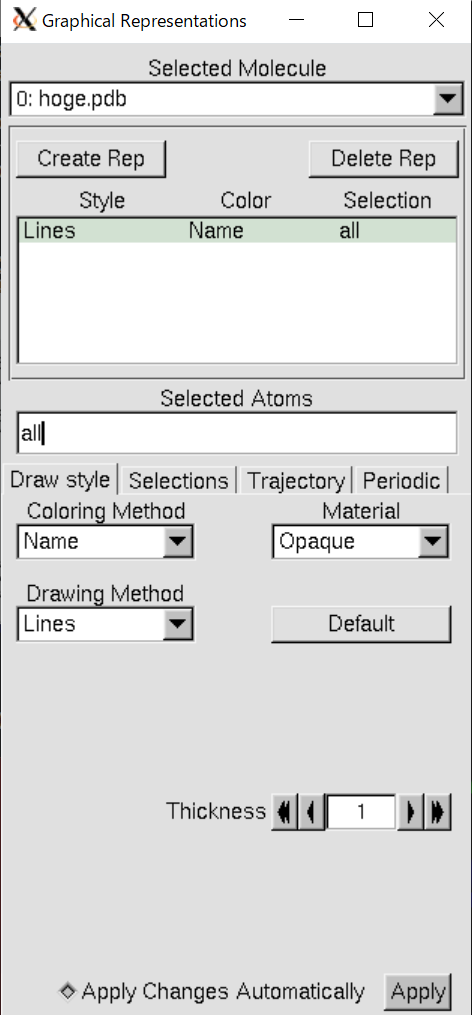
しかしこれだけではただ線が表示されているだけであり、はっきり言って見にくい。そのためグラフィックを変更して自分の見やすいように調整してみよう。グラフィックを変更する場合はGraphicsタブを押す。すると次のようなGUIが出現する。
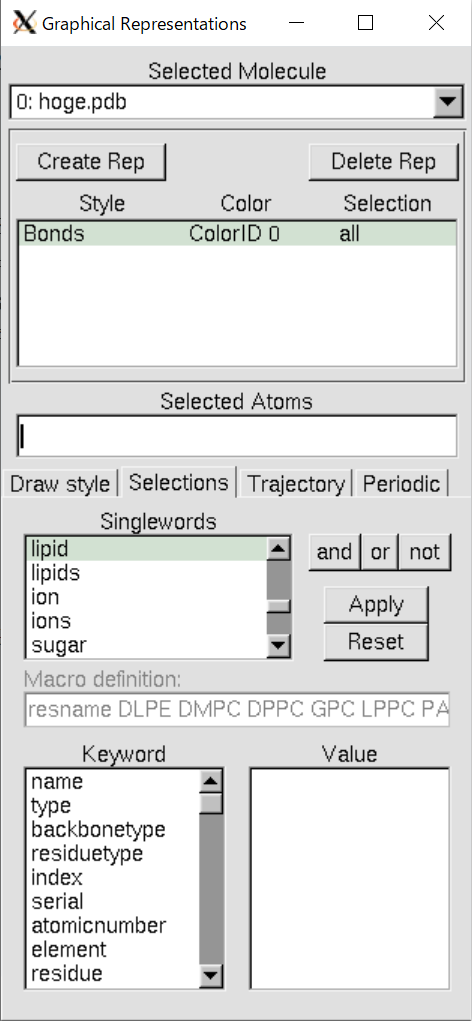
この"Graphical Representations"のGUIはVMDを触る上でマスターすべき最初の難関である。慣れてしまえばなんてことはないが、初めはこのGUIの見方が分からない。そのため使い方を解説していく。
Rep¶
画像中央の緑色のついた"Lines Name all"と書かれたものが、Repと呼ばれるものである。
これはどんな描写形式でトラジェクトリを再生するかを選ぶためのものである。このRepは"Create Rep"をクリックすることでさらに追加できる。すなわち描写形式をいくつも作っておけるということである。
反対に消したいときは消したいRepをクリックした後に"Delete Rep"をクリックすることで消すことができる。
また消したくはないがこの描写形式をやめたいという時はやめたいRepをダブルクリックすることでそのRepの描写を非表示にすることができる。
以下でRepのStyle, Color, Selectionについて述べる。
Style¶
分子や原子をどんな表示形式にするかを選ぶことができる。表示形式の変更はDrawing Methodの下矢印マークをクリックすることで変えられる。以下主要なものについて簡単に記述しておく。
Lines: 極めて細い線で結合を表示し、結合の種類によって色分けが行われる(後述の"Color"を"name"にしている場合)。もっとも動作が軽いstyleであるため、容量の大きいトラジェクトリを再生するのに役立つ。
Bonds: Linesと色分けは同じであるが、結合が太く分子模型のように描写される。結合に関して見たい時はこのstyleで見たらいいと思われる。
Hbonds: 少しマニアックかもしれないが、水素結合を見ることができる。と言っても水素結合はトポロジーに記述されている共有結合ではないため単純にH-OやH-Nの原子間距離と、角度が定義の範囲を満たしていれば結合として表示される。定義の範囲はそれぞれ"Distance Cutoff"と"Angle Cutoff"を変えることで変更できる。デフォルトで適切と思われる値になっているが、H-Nといった特定の水素結合に着目する場合はH-Nの水素結合距離を文献やネットで調べてその値に設定するとよい。
VDW: 最も使うsytleの1つである。分子を全て粒子として表示し、結合は表示しない。VDWによるRepとBondsなどのRepを組み合わせて使用することも多い。これにより原子が飛ぶといった計算が走らない時の要因を特定することができる。また水やイオンをBondsなどで表示するとかえって分かりづらいためVDWで表示して振る舞いを見ることが多い。
CPK: 未だに何の略か分からない。しかしこの描写が一番実際の分子を程よく表していると個人的には思っている。VDWにすると粒子同士が重なって見にくいことがある。VDWで表示する粒子半径を小さくすればよいだけではあるが、そんなことをしなくてもCPKにすると原子と結合が非常に見やすい。
Licorice: このstyleはT先輩リスペクトである。正直Bondsとほとんど同じである。但し、Licoriceの方がなんか見栄えがよく見える。そのため筆者は脂質分子をこのstyleにすることが多い。
NewCartoon: タンパク質を研究している人にとっては必須のstyleである。Cartoonよりもこちらのstyleを使う。二次構造がある場合にはα-ヘリックスまたはβシートでの表示がなされる。発表などではまずこのstyleを使うであろう。そうでないとBondsなどで表示してもタンパク質かどうかすら見ている人には分からない。但し逆に自分で分子の振る舞いを見る場合、NewCartoonでは残基や原子など本来の姿を見えなくしてしまうため注意が必要である。特に残基間の相互作用が気になる場合は個人的にはCPKがおすすめ。
QuickSurf: このstyleも割と発表などで使用する。分子などを粗く見ることで液体のように表示できる。そのため水分子をQuickSurfで表示すると、本物の水かのように見せられる。詳しくはT先輩のサイトにやり方が書いてあるのでそちらを参考にしてほしい。ここでは割愛する。
Color¶
そのままの意味で分子の色を変えることができる。こちらも色々種類があるが、主に使用するのはnameとColorIDである。nameでは原子の種類ごとに自動で色分けがなされる。基本的にはこれで問題ないが、場合によっては見にくいため色を個別に変えたい場合がある。その際にはColorIDを使用する。ColorIDを選択すると隣に番号が表示される。この番号は各色の種類を示しており、32番まである。
ぜひ自分好みの色を探してみてほしい。
Selection¶
"Style"も種類が豊富であったが、それ以上に奥が深いのがこのSelectionであろう。このSelectionでは表示したい分子の種類や原子の種類を選べるだけでなく、特定の座標の半径何Å以内の原子といった非常に様々な指定の仕方がある。それらを指定したい場合にはSelected Atomsの欄にキーワードを打ち込めばよい。例えば先ほどの写真では"all"と打ち込まれており、これは全ての原子を表示できる。
キーワードには主に以下のものがある。
all: 全ての原子を表示
protein: タンパク質を表示
water: 水を表示
lipid: 脂質を表示
ion: イオンを表示
※注意 lipidとしても表示されない脂質種もある。例えばPOPEは表示されるがPOPGは表示されないので注意が必要である。ionなどのキーワードにも同様に当てはまる。
他にも多数のキーワードがあり、どんなキーワードがあるか知りたい場合はSelectionsのタブを押して”Singlewords”の一覧を見るとよい。
キーワードには先に述べたものとは別に、キーワードと番号や名前を組み合わせて指定するものもある。これは画像下部の"Keyword"の一覧に表示されているものがそうである。具体的な一例を以下に挙げておく。
name CA
resname GLY
index 27
Chain A
簡単に説明する。
nameは原子のタイプ名の検索が可能である。上記の例ではCAというタイプの原子のみを選択している。
resnameは残基名の検索であり、GLYやALAといった残基名と組み合わせて使う。またタンパク質でなくとも脂質や水、イオンといった分子種を選ぶ場合にも使用する。
indexは全ての原子に対して0番から順番に番号を振った時の番号である。似たような機能にserialがあるがこちらは1番始まりである点で異なる。
ChainはpdbなどでChainIDの記述がある場合に選べるが、タンパク質を扱わない人には無縁かもしれない。
キーワードは分かったがそもそもどんな原子タイプや残基名が使用可能か分からないという人は安心してほしい。上の写真の"Keyword"の中から選びたいキーワードをクリックすると、そのキーワードに対して使用可能な名前や数字が"Value"の方に表示される。ちなみにこの"Value"はVMDに元々組み込まれているものではなく、pdbやgroをVMDで読み込んだ際に原子タイプや残基名も読み取られ、それが"Value"に表示されている。
さらに勘のいい人は上の画像を見て"Singlewords"のカラクリにも気づいたかもしれない。画像ではlipidという単語を選択しているが、その際に"Macro definition"にresname DLPE DMPC...などと書かれていることに気づいただろうか?
そうlipidというキーワードはresname DLPE DMPC...と選択するのと全く同義である。ちなみに"Singlewords"でキーワードをダブルクリックするとそのキーワードの定義がSelected Atomsに打ち込まれる。lipidをダブルクリックした場合には次のように打ち込まれる。
resname DLPE DMPC DPPC GPC LPPC PALM PC PGCL POPC POPE
このためこの中にあるPOPCやPOPEはlipidとするだけで選択ができる。反対にPOPGやPOPSといったものはないため、選択ができない。これが「lipidとしても表示されない脂質種もある。」の理由である。
おまけ¶
"Draw style"のタブの中に"Colorring Method"や"Drawing Method"以外に"Material"の項目がある。
これは名前の通り分子の材質を変えることができる。かなり癖の強いものが色々ある。基本的にデフォルトの"Opaque"で良いと思うがT先輩のオススメは"AOshiny"である。少し明るいが影がついて立体感が出る。
ということでAOshinyを使いましょう。あとはどうしても分子がごちゃごちゃして内部が見にくいということがあるので必要に応じてTransparentで透明にするものアリ。"Material"も色々触ってみてください。
初級編まとめ¶
初級編からかなり長くなったが、まだまだ紹介すべきことはたくさんある。そのため次のページでは中級編として紹介する。またここで紹介した以外にもStyleやColor、Selectionは様々な種類やキーワードがあるのでそれらを色々試してみてほしい。