高分子鎖のからみあい点間分子量(からみあい)調整¶
からみあいとは¶
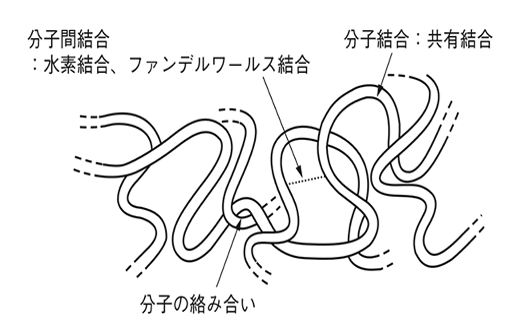
端的に説明すると以下の図のように分子鎖同士がフックしあっている点間の距離のことを指す。この物性値は高分子の歪みに対する応力を示す因子の一つであり、タイムスケールが小さいMD計算では実験値に収束しないため作為的に調整する必要がある。からみあいの調整を行なっているプログラムの内容は各自確認して欲しいが、高分子鎖を構成するモノマー重心間のなす角度を認識させて、それぞれのモノマーに対してからみあいの値が実験値と一致するように単位ベクトルを与えることを繰り返すことでプリミティブパス(高分子鎖末端の2点間をからみあい点を経由して繋いだ最小距離)を調整する。
からみあいを変化させたトラジェクトリファイルを生成する。¶
からみあいを調整するためにこのトラジェクトリファイルを生成する段階でスパコンを利用する。手元のパソコンからスパコンに以下のようにrsyncすることで様々な実行プログラムが整理されたディレクトリとからみあいを調整するディレクトリを持ってくる。
# からみあいを調整するディレクトリをpolymerparticleというディレクトリに入れる場合
rsync -avh /mnt/ImPACT02/temp_yoshida/AdjustEntanglement RCCS:/home/users/dfw/2022/polymerparticle/
#実行プログラムをまとめられたディレクトリをスパコンに入れる場合
rsync -avh /mnt/ImPACT02/temp_yoshida/bin RCCS:/home/users/dfw/2022/
次に実行ファイルを動かしていく。手元のパソコンにおいてもスパコンにおいてもプログラムを動かすためにはコンパイルという作業を行う必要がある。実行ファイルが存在する~/bin/AdjustEntanglement/に移動し、Makefileに従ってadjustentanglement.cppというファイルをコンパイルをしていく。この際Makefileには筆者の環境での内容が記載されているため、このファイルを自分の環境用に書き換える必要がある。具体的には以下の部分を自分の環境用に書き換えて欲しい。
MKLROOT=/local/apl/lx/intel/
GROMACS=/local/apl/lx/gromacs2018/
QHULL=/home/users/dfw/2022/apl/liblbfgs-1.10/install/
LIBLBFGS=/home/users/dfw/2022/apl/netcdf-3.6.3/install/
NETCDF=/home/users/dfw/2022/apl/netcdf-3.6.3/install/
MYLIBRARY=/home/users/dfw/2022/bin/library
INCLUDE = -I$(MKLROOT)...
-I$(NETCDF)...
...
LIBDIR = -L$(MKLROOT)...
-L$(NETCDF)...
...
基本的にはINCLUDEやLIBDIRの部分は変えなくて良いかもしれない。主に/home/user/dfw/2022/で始まる部分を自分のディレクトリ上のPATHに変更して欲しい。また筆者はGROMACSのバージョンは2018を使っていたが/local/apl/lx/にある自分の使っているバージョンのものに書き換えて欲しい(新しいものの方がいいかも)。この後にmakeでコンパイルが通れば成功である。
しかし、上記のMYLIBRARYに存在するプログラムを参照しているためこのプログラムも自分のパソコンの環境用にコンパイルする必要がある。コンパイルする前にmake cleanを打ち込んでコンパイルする前の状態に戻してから上記と同じようにMakefileを書き換えた上でmakeを行なってコンパイルしてみよう。ls -ltrhでlibmd.aが生成されていることを確認できたらコンパイル成功である。
これでもう一度binの中にあるAdjustEntanglementに戻ってmakeを実行して欲しい。make時にエラーが出た時は大抵ファイルが見つからない系の内容だと思われるのでfindコマンドなどを用いてファイルのPATHを特定してMakefileを書き換えて欲しい。それでもエラーが直らない場合は研究室の先輩などに聞いてみよう。実行ファイル(adjustmententanglement_3.7)ができたらldd adjustmententanglement_3.7で実行ファイルが実行できるか確認して欲しい。not foundというものが表示された場合、必要に応じて~/.bashrcの中のPATHを通すようにしよう。
次にbinディレクトリから出て、AdjustEntanglementのディレクトリに移動してからみあいを調整したい構造ファイル(~.gro)を持ってくる。その後Run.shを実行することでからみあいを変化させたトラジェクトリファイルを生成する。実際にプログラムを動かす場合は~.inputの内容を書き換える必要がある。自身の計算条件に応じて書き換えて欲しい、特にスパコンの環境に合わせてCORE数の指定は確実に行おう。Run.shファイルの中身は以下のようである。
/home/users/dfw/2022/bin/AdjustEntanglement/adjustentanglement_3.7 -i adjustentanglement.input
トラジェクトリファイルからからみあい点間分子量を出力させる。¶
この過程は上記の過程で生成したトラジェクトリファイルの各フレームにおけるからみあい点間分子量を数値として出力するものであり、手元のパソコンで行う。手元のパソコンのbin内にあるZ1_input内にあるmakefileの中身を書き換えてmakeを実行してコンパイルを行う。makefileの書き換えは上記と同じように行なってもらいたい。特に/home/yoshida/で始まる部分は自分の環境に合わせて書き換える。findやlddを駆使してプログラムに必要なファイルに対するPATHを通して欲しい。
AdjustEntanglementの中にあるZ1_inputに入り、z1_input.inputの内容を自身の計算条件に応じて書き換えて欲しい(主にInputのトポロジーファイルとトラジェクトリファイル)。その後、binの中にあるZ1_inputの実行ファイルを以下のように実行する。
$/home/(ユーザー名)/bin/Z1_input/z1_input_1.2 -i z1_input.input
調整したい物性値(からみあい)を持つ座標ファイルの構造を取り出す。¶
Z1_inputの実行ファイルを実行したら、Z1_input内のZ1に入ってloop.shを実行する(この時loop.sh内の/home/yoshida/で始まる部分を自分用に書き換える)。
$./loop.sh
loop.shを実行後にNe.logをviで開いてからみあい点間分子量が適した値をもつフレームを探す。 その後AdjustEntanglement(binの中のやつではない)のディレクトリまで戻り、vmdで初期構造ファイル(~.gro)と先ほど生成したトラジェクトリファイル(~.dcd)を選択し、上記で探したフレームの構造ファイルを取り出す。