1. 系の作成¶
任意のMDシミュレーションを開始するためには、まず大前提として系が用意されている必要がある。 ここでは直鎖アルコールの構造を作成し、それをボックス内に配置することで、アルコール分子の二重膜が含まれる系の作成をしてみたいと思う。
なお、今回は系の作成を簡単にするために、自動で構造を生成してくれる外部サイトを利用する。 インターネット環境が必要となるので、通信環境の良いところで取り組んでほしい。
アルコール分子の作成(CHARMM-GUI)
アルコール二重膜系の作成
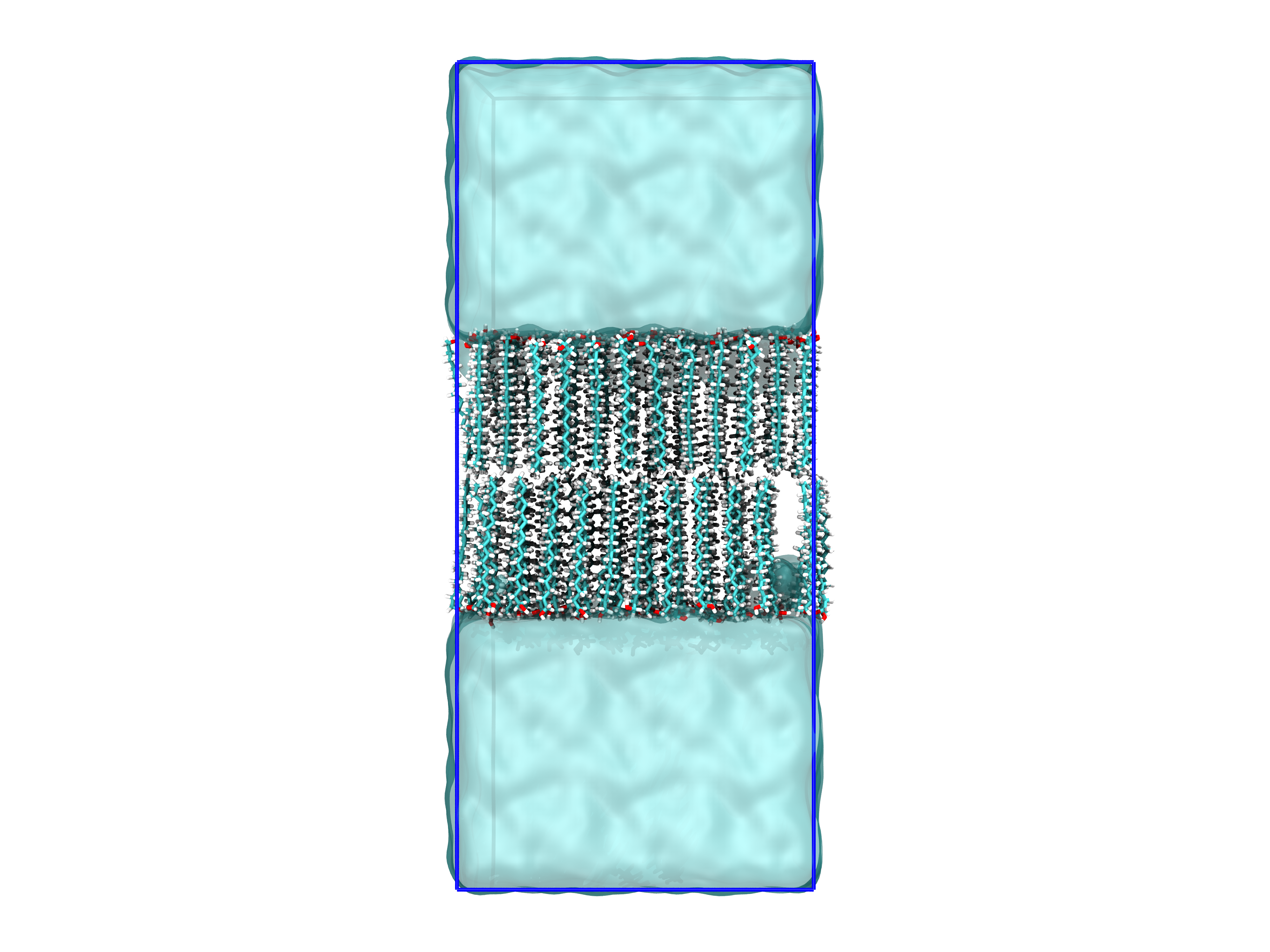
今回作成する系は図のようになる。 アルコール分子が整列して二重膜を形成し、その両側にはバルクの水が存在するような系を作成する。
アルコール分子の作成(CHARMM-GUI)¶
まず、シミュレーションする系に含まれる分子であるアルコール分子の構造を作成する。 簡単な分子構造を作成し、構造ファイルを書き出すにはCHARMM-GUIが便利である。 CHARMM-GUIはWeb上で動くプログラムであり、PDBファイルの作成からシミュレーションのインプットファイルの作成まで、多くの便利な機能が備わっている。
注意
最近のアップデートにより、CHARMM-GUIの利用にはメールアドレスとパスワードによるログインが必要となった。アカウント作成ページで名前・所属機関・メールアドレスの登録をして利用しよう。

今回はCHARMM-GUIのトップページから「Input Generator」を選択し、その中の「Ligand Reader and Modeler」を利用する。 一番下までスクロールすると、次のような画面が現れるはずである。
この画面の枠内では分子を描画することができる。今回はアルコール分子として、C16H33OHの構造式を描いてみることにする。
枠の左側のツールを用いて直鎖アルカンを描画し、右側のツールで元素を変更する。 分子の骨格ができたら必ず枠上のHマークから分子に水素を付加しておくこと。 これを忘れると後にエラーとなってしまう。 最終的に下図のようになればOKである。
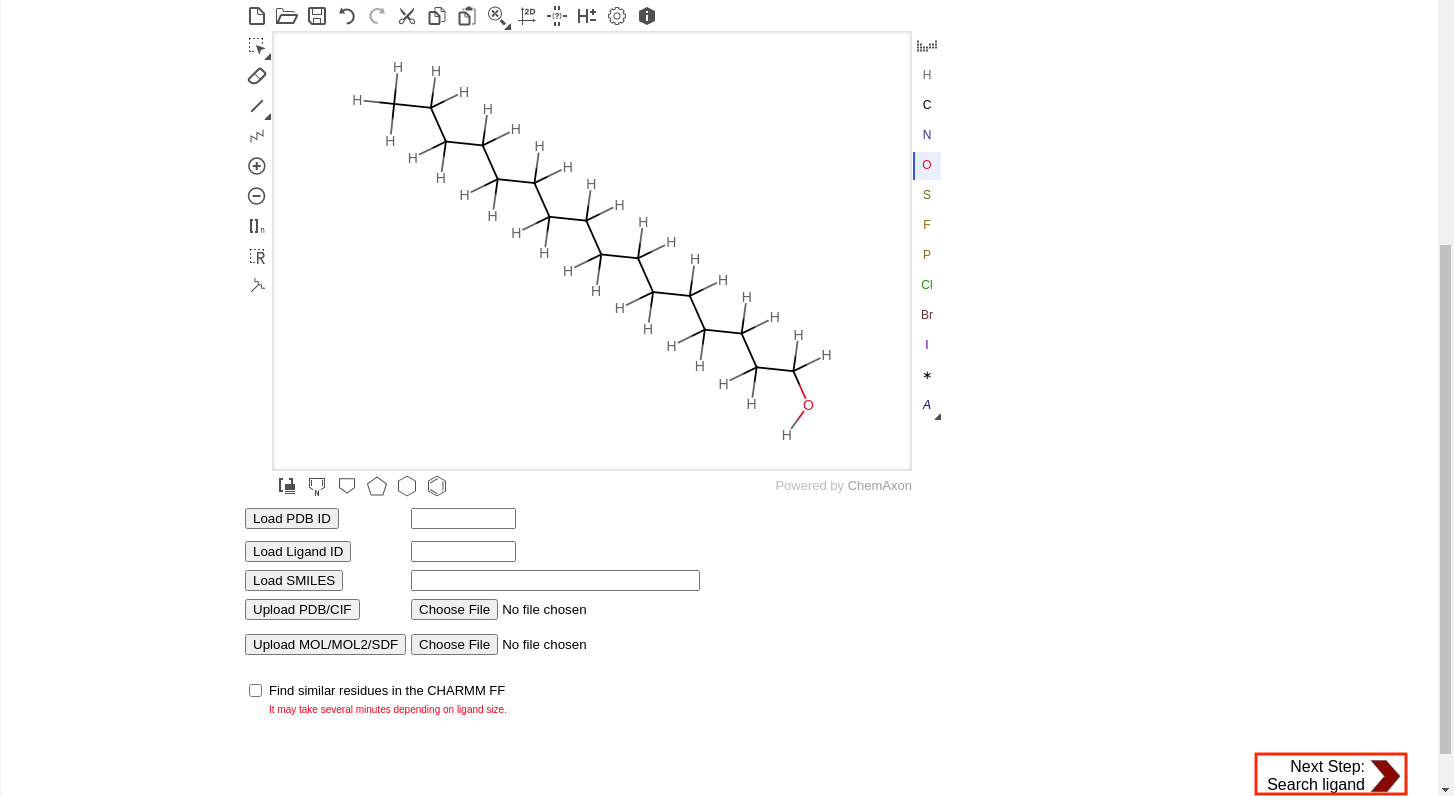
一番下の「Next Step: Search ligand」を押すと、次の画面に進む。 作成した分子と一致する構造がCHARMM-GUI上にあった場合や、似たような構造があった場合はここで表示される。 一致するものがなくても、その分子にオリジナルの名前(VMDではresnameで分類される)をつけることができる。 今回は「LAL」(Linear-chain Alcohol Ligand)という名前をつけておくことにする。
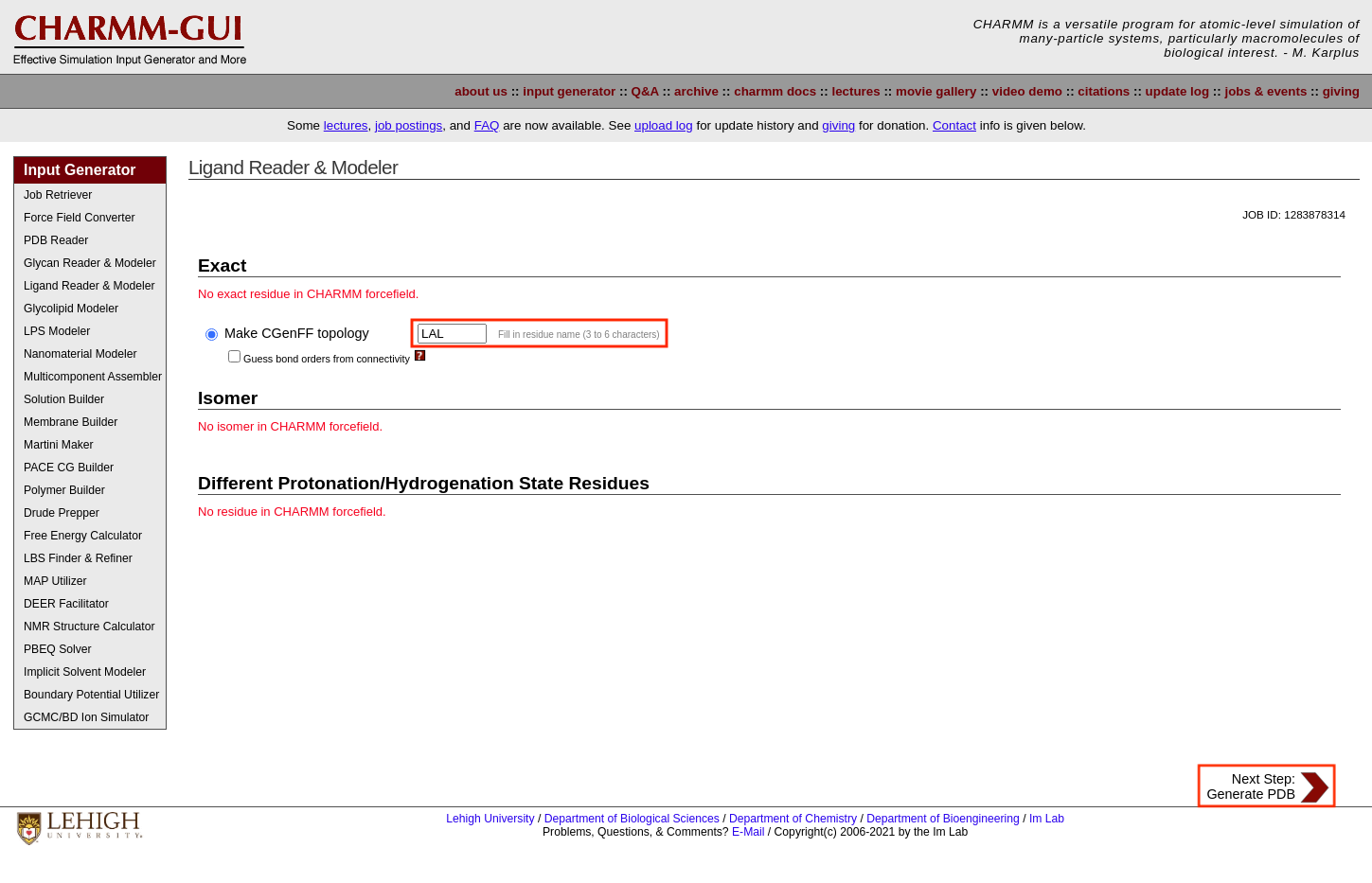
「Next Step: Generate PDB」を押すと、プログラムの処理が始まる。 しばらくすると次のページへ移動し、PDBファイルやpsfファイル、インプットファイルなどが生成する。 これらは「download.tgz」から一括でダウンロードしておくとよい。
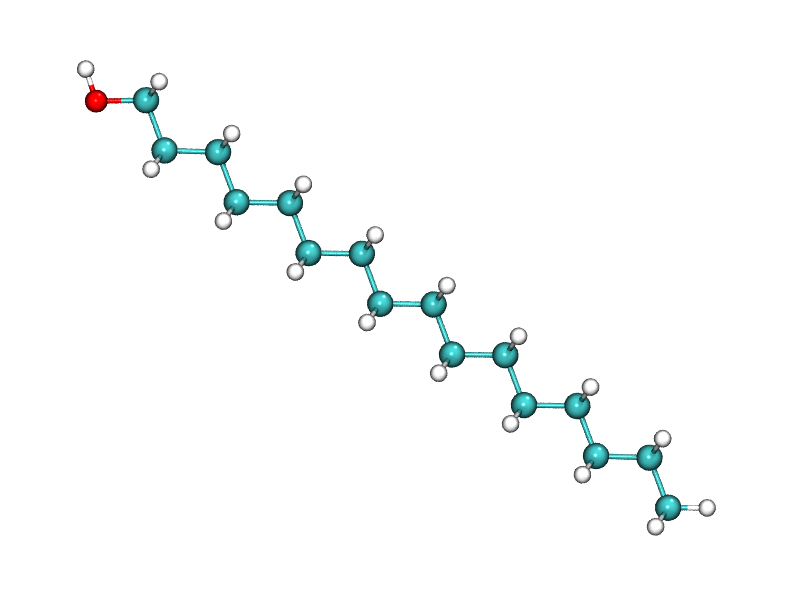
ダウンロードしたファイルを展開し、PDBファイルをVMDで確認すると、アルコール分子が正しく作成できていることがわかる。
アルコール二重膜系の作成¶
ここまでで、系の構築に必要なアルコール分子の構造を作成することができた。 二重膜系を作成するためにはPACKMOLなどを使用して分子を並べてもよいが、今回は便利な二重膜系作成ツールがあったのでそちらを利用する。
MemGenにアクセスし、先ほど作成したアルコールのPDBファイルをアップロードする。 オプションでは膜中の分子密度や水の量などを指定できるので、今回は次のように設定する。
| オプション | 値 |
|---|---|
| Lipids per monolayer | 128 |
| Water molecules per lipid | 30 |
| Add salt (mMolar) | 0 |
| Area per lipid (Å2) | 20 |
設定が完了したら「Submit」を押すと、プログラムが動いて二重膜の構造を自動生成してくれる。 二重膜系のPDBファイルを「Download Membrane PDB」からダウンロードしておこう。
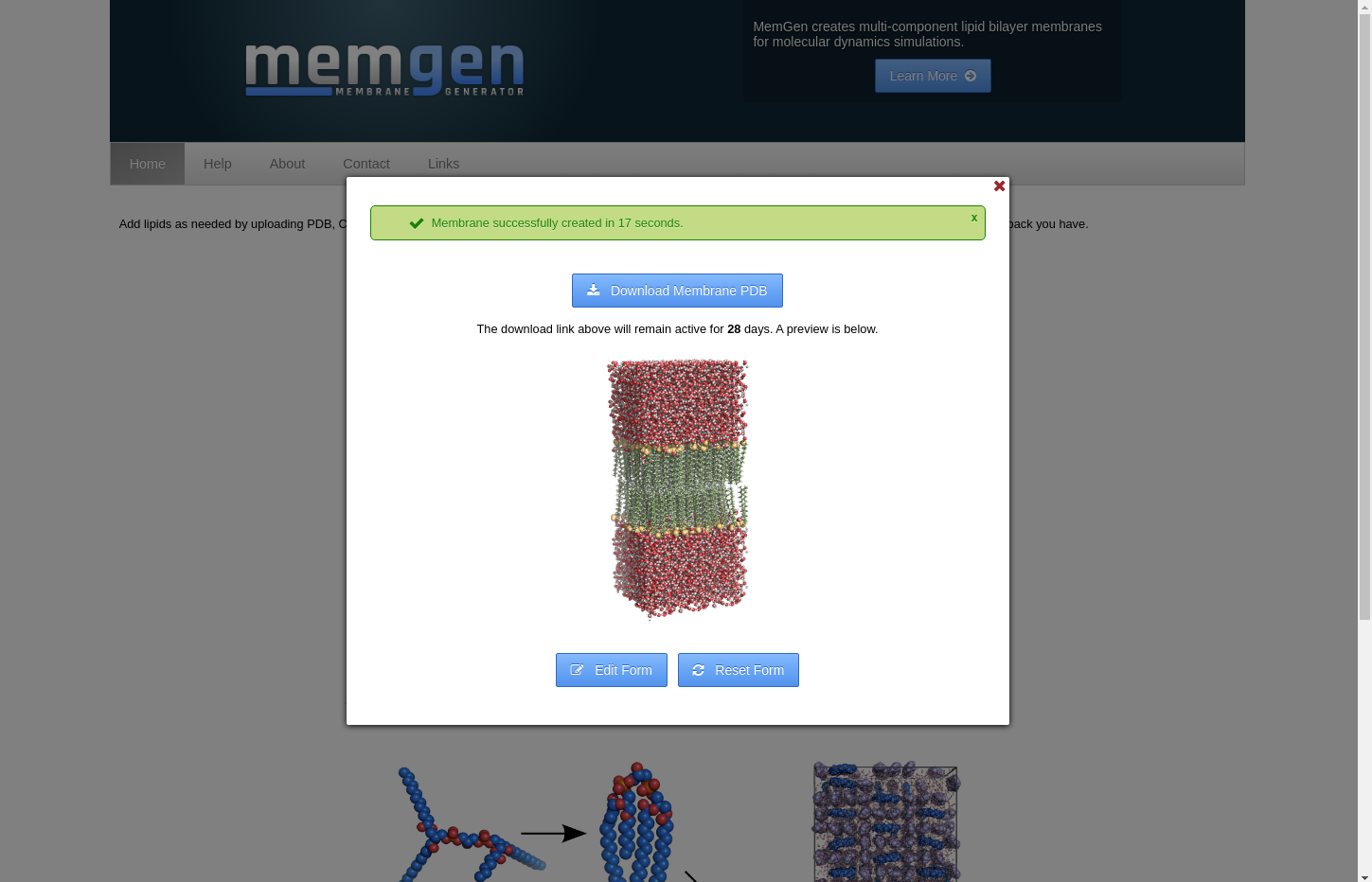
ダウンロードしたPDBファイルの名前をsystem.pdbに変更し、テキストエディターで内容を確認する。 今回は(多少数値は前後するであろうが)アルコール分子が256個、水分子が7680個含まれる系で、ボックスサイズは(x, y, z)=(53.666, 53.666, 124.570)であることがわかる。
また、その次の行からは原子の種類と座標が記述されている。 詳しくは調べてほしいが、PDBファイルの形式は単なるスペース区切りではなく、何文字目がどの情報を表すかが厳密に決まっている。 GROMACSでPDB形式を扱うことはほぼないが、NAMD等を使用する際には必要となるので覚えておくとよい。
system.pdb
REMARK
REMARK MEMBRANE PATCH GENERATED BY WEBSERVER MEMGEN: http://memgen.uni-goettingen.de
REMARK
REMARK PLEASE CITE:
REMARK
REMARK Christopher J. Knight and Jochen S. Hub
REMARK MemGen: A general web server for the setup of lipid membrane simulation systems
REMARK Bioinformatics, 31:2897-2899 (2015)
REMARK
REMARK THIS PDB FILE CONTAINS: LAL 256
REMARK Water 7680
REMARK Na+ 0
REMARK Cl- 0
REMARK
CRYST1 53.666 53.666 124.570 90.00 90.00 90.00 P 1 1
MODEL 1
ATOM 1 C1 LAL 1 2.640 54.580 63.460 1.00 0.00 C
ATOM 2 C2 LAL 1 2.610 53.670 64.700 1.00 0.00 C
ATOM 3 C3 LAL 1 2.670 54.470 66.010 1.00 0.00 C
ATOM 4 C4 LAL 1 2.760 53.530 67.220 1.00 0.00 C
ATOM 5 C5 LAL 1 3.190 54.280 68.480 1.00 0.00 C
ATOM 6 C6 LAL 1 3.150 53.230 69.610 1.00 0.00 C
ATOM 7 C7 LAL 1 3.450 53.890 70.960 1.00 0.00 C
ATOM 8 C8 LAL 1 3.120 53.000 72.170 1.00 0.00 C
ATOM 9 C9 LAL 1 3.290 53.950 73.360 1.00 0.00 C
同時にVMDでも系の構造を確認しておく。

まとめ¶
ここまで、次の手順でアルコール二重膜系の構造を作成した。
CHARMM-GUIによりアルコール分子の構造を作成した。作成したアルコール分子を用いて
MemGenで二重膜系を作成した。
しかし、このアルコール分子はオリジナルの分子であるため、GROMACSにこの分子を認識させる必要がある。 次回はその点を含め、作成した系をGROMACSで扱う方法を見ていく。